
فهرست مطالب
مقدمه
امروزه تعیین توالی DNA به یکی از مهمترین تکنیکهای موجود در علم زیستشناسی تبدیل شدهاست. در این روش ترتیب قرارگرفتن نوکلئوتیدها در یک قطعه از DNA مشخص میشود. توالییابی که در زمان گذشته به صورت سنتی مورد استفاده قرار میگرفت؛ بسیار زمانبر و گران بود و پاسخگوی تحقیقات گسترده دانشمندان را نمیداد. در نتیجه تکنولوژیهای عملکردی بالا برای تولید داده یا نسل جدید توالییابی گسترش یافت که قادر به توالییابی هزاران یا میلیونها توالی به صورت همزمان میباشد. در دهههای اخیر تعیین توالی DNA در زمینه پزشکی، کشاورزی و دیگر زمینههای علوم زیستی از جایگاه ویژهای برخوردار بودهاست.
تاریخچه توالی یابی
اولین تلاش برای توالییابی بر روی rRNA و tRNA میکروبی و همچنین RNAهای تکرشتهای باکتریوفاژها متمرکز شد. علت آن هم این بود که RNAها تکرشتهای و کوتاهتر هستند و پیچیدگی آنها کمتر از DNA یوکاریوت است. همچنین آنزیمهای RNAse قادر بودند تا زنجیره RNA را برش دهند.
در نهایت در سال ۱۹۶۵ روبرت هالی و همکارانش موفق شدند اولین توالی نوکلئیک اسید مربوط به tRNA حمل کننده آلانین را در مخمر شناسایی کنند. همزمان با آنها فردی به نام سنگر و همکارانش تکنیکی مبتنی بر شناسایی قطعات رادیولوژیشده و حذفشده بعد از جداسازی دوبعدی پایه ریزی کردند. پس از آنها محققان تلاش خود را بیشتر کردند
تا اینکه در سال ۱۹۷۷ پیشرفتی بزرگ برای همیشه تکنولوژی تعیین توالی را تغییر داد و روش خاتمه زنجیره یا دی دئوکسی معرفی شد. توالییابی از آن زمان تا امروز دچار تغییر و تحولهای چشمگیری شد و چندین روش توالییابی که هریک مزایایی نسبت به دیگری دارند معرفی شدند.
انواع روشهای تعیین توالی
توالییابی نسل اول
- روش سنگر
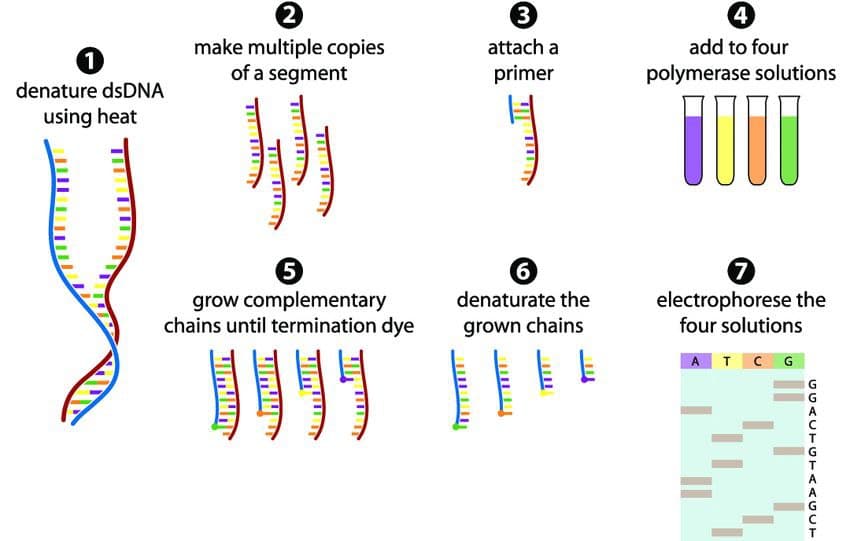
این روش ابزاری برای رمزگشایی ژنوم فراهم کرد. در سال ۲۰۰۱ حدود ۹۰ درصد از ژنوم انسان با استفاده از روش توالییابی اتوماتیک سنگر تعیین توالی شد. در این روش برای تهیه الگو ممکن است از یک مولکول کامل DNA یا از الگوهای تکثیر شده به صورت کلون، از یک مولکول منفرد استفاده شود.
مبنای این تکنیک استفاده از آنالوگهای شیمیایی نوکلئوتیدی به نام ddNTPs یا همان dideoxynucleotids است. این مولکولها فاقد گروه OH بر روی کربن 3 خود برای گسترش زنجیره DNA هستند و نمی توانند با ۵ فسفات dNTP بعدی خود واکنش دهند؛ بنابراین هنگامی که در زنجیره در حال تکثیر اسید نوکلئیک قرار گیرند واکنش متوقف خواهدشد. ترکیب ddNTP رادیو لیبل شده همراه با غلظتهای مشخص از ddNTP استاندارد در واکنش سنتز DNA منجر به تولید رشتههای DNA با طولهای مختلف میشود.
مواد مورد نیاز سنتز DNA به ۴ لوله آزمایش که هر کدام حاوی یک نوع باز ddNTP هست انتقال داده میشوند. dNTPها به طور تصادفی توسط آنزیم DNA پلیمراز به رشته در حال ساخت اضافه میشود. نهایتاً در هر لوله آزمایش قطعاتی با طول مختلف تولید میشود که قطعات تولید شده بر روی ژل پلی اکریل آمید قرار میگیرند. سهولت در روش سنگر باعث شدهاست که این تکنیک برای سالهای زیادی مورد استفاده قرار گیرد.
استفاده از نوکلئوتیدهای فلورسنت از جمله پیشرفتهای اخیر در این روش است که باعث میشود به جای ۴ لوله آزمایش واکنش تنها در یک لوله آزمایش انجام شود. هر یک از قطعات DNA با یک رنگ فلورسنت نشانهگذاری میشوند. بنابراین هر قطعه DNA دارای اندازه و رنگ منحصربهفردی خواهد بود. همچنین استفاده از الکتروفورز لوله مویین از دیگر روشهای توسعه یافته است. این دو پیشرفت باعث توسعه ماشینهای توالی یابی خودکار شد و به آنها در این زمینه کمک کرد.
- روش ماکسام_گیلبرت

در توالییابی به روش ماکسام_گیلبرت مبنا هضم شیمیایی است که در سال ۱۹۹۶-۱۹۹۷ ابداع شد. در این روش نیاز به پلیمرازیسیون قطعات DNA نیست. روش توالییابی به اینصورت است که در مرحله اول برای آمادهسازی نمونه مولکول DNA را دناتوره میکنند تا به صورت تکرشتهای درآید. پس از آن در انتهای ‘۵ برچسبگذاری انجام میدهند که در این روش اغلب از فسفر ۳۲ استفاده میشود.
در مرحله بعدی یعنی در مرحله دوم توسط پیپریدین و دو ترکیب شیمیایی دیگر که به پورینها و پیریمیدینهای مولکول DNA حمله میکنند، استفاده میشود. هر کدام از ترکیبات شیمیایی مولکول DNA را در محل یکی از چهار باز آدنین، تیمین، گوانین و سیتوزین برش میدهد. با قرار دادن چهار لوله آزمایش برای هر ترکیب شیمیایی میتوان چهار نمونه از قطعات با برشهای یکسان را به دست آورد بعد از اینکه قطعات برش خورده DNA به دست آمد؛
هر کدام از آنها بر روی ژل پلی آکریل آمید با درصد بالا مورد الکتروفورز قرار میگیرند. برای تمایز بین قطعات مختلف از برچسبهای رادیو اکتیو استفاده میکنند. برای خوانش DNA هم باید از کوچکترین قطعه در پایین ژل شروع کرد. برای نمایش و مرئیسازی بخشهای مورد نیاز، ژل مورد نظر در معرض اشعه X قرار میگیرد تا اتورادیوگرافی انجام شود و نوارهای تیرهای را که بیانگر مکان مولکول رادیواکتیو شدهاست نشان بدهد. از حضور و غیاب قسمتها میتوان توالی رشته را استنباط کرد.
گسترش روشهایی مانند واکنش زنجیرهای پلیمراز و فناوری DNA نوترکیب با فراهم آوردن مقدار قابل توجهی DNA که ماده اولیه برای توالییابی محسوب میشود؛ انقلابی در ژنومیکس ایجاد کرد.
توالی یابی نسل دوم
- روش توالییابی Pyrosequencing

از مزایای این روش میتوان به استفاده از دئوکسی نوکلئوتیدهای طبیعی و عدم استفاده از الکتروفورز که فرآیند طولانی و زمانبر محسوب میشود؛ اشاره کرد. در این روش از PCR جهت ازدیاد قطعات مورد نظر استفاده میکنند و این امر باعث افزایش سرعت و بازدهی توالییابی DNA میشود.
این روش در سال ۲۰۰۵ توسط شرکت Life Sciences ابداع و بعدها توسط شرکت Roche خریداری شد. همچنین توانست به اولین تکنولوژی تجاری تحت عنوان NGS تبدیل شود. از آن پس از اصطلاحات NGS جهت توصیف مجموعهای از فناوریها با قدرت بالاتر نسبت به توالییابی سنگر مورد استفاده قرار گرفت. این تکنولوژی تجاری قادر به توالییابی تعداد زیادی از قطعات DNA در یک ران میباشد
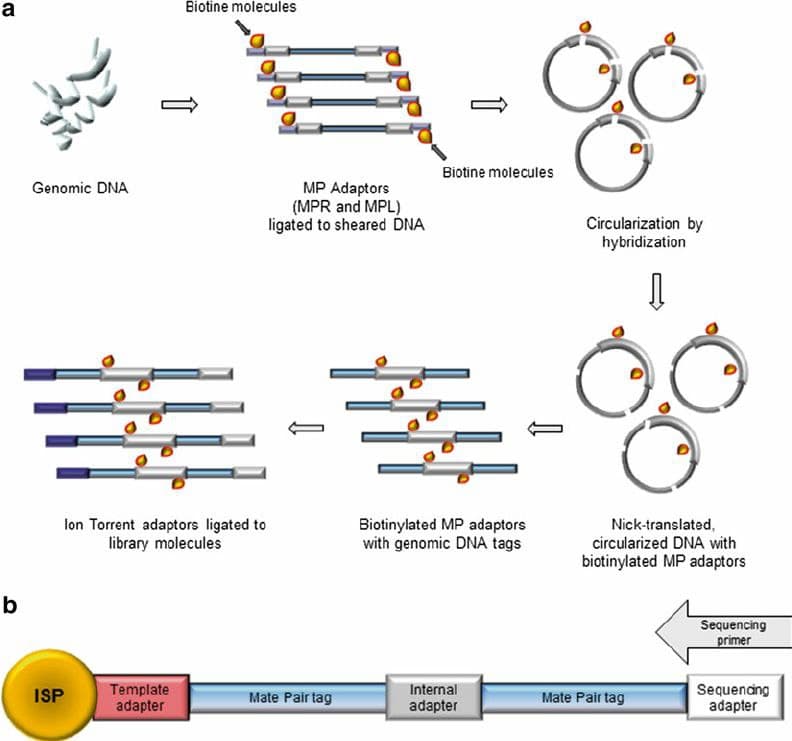
در مرحله اول DNA به صورت قطعات ۳۰۰ تا ۵۰۰ بازی شکستهشده و سپس با استفاده از آداپتور به بیدهای فلزی متصل میشود. در این روش آداپتور نقش مهم را بازی میکند؛ زیرا آداپتور در انتهای ‘۵ خود دارای گروه بیوتین و بیدهای فلزی با استروپتوآویدین پوشیده شدهاست. اولین نقش آداپتور اتصال قطعات DNA تکرشتهای به بیدهای فلزی کوچک است. در واقع اتصال به واسطه تمایل بالای استرپتوآویدین به بیوتین انجام میگیرد.
آزمایش به گونهای طراحی شدهاست که به هربید یک قطعه DNA تکرشتهای متصل میشود. در مرحله بعدی هر بید در یک قطره آب در امولسیون روغن قرار میگیرد و واکنش زنجیرهای پلیمراز آب در امولسیون روغن انجام میشود. سپس واکنش PCR جهت تکثیر قطعات DNA انجام میشود. نقش دوم آداپتور فراهم کردن جایگاه برای اتصال پرایمر و تکثیر قطعه مد نظر است. از آنجا که این تکنیک نیازمند DNA الگوی تکرار شدهای میباشد پس از پایان مرحله، محصولات PCR دناتوره میشوند. پس بدین ترتیب از تکثیر بر روی هر دانه در حدود یک میلیون قطعه DNA تکرشتهای جهت تعیین توالی ایجاد میشود.
هدف از انجام مراحل PCR افزایش تعداد قطعه هدف است تا سرعت عمل برای توالییابی افزایش یابد. در مرحله بعدی امولسیون شکسته میشود و به محتوای درون آب چاهکهای پیکولیتری در سطح یک پلیت منتقل میشوند. در داخل هر چاهک تنها یک بید قرار میگیرد. سپس آنزیمهای مورد نیاز شامل سولفرولیراز، لوسیفراز، آپیلاز به همراه سایر مواد مورد نیاز برای تکثیر به درون چاهکها انتقال داده میشوند. به این ترتیب پایروسکوئنسینگ در هر چاهک انجام میگیرد. در این تکنیکها میتوان بیش از یک میلیون نمونه را به طور همزمان تعیین توالی کرد.
اولین ماشین توالییابی با مقیاس بزرگ که بهصورت گسترده توسط مشتریان استفاده شد GS20 نام داشت که بعدها به Gas FLX۴۵۴ با تعداد بیشتری چاهک در پلیت پیکوتیتر ارتقا یافت؛ که سرعت، وضوح و کیفیت بالاتری داشت. همچنین همزمان با کاربرد موفقیت آمیز تکنیکهای دیگری به وجود آمده که مهمترین آنها روش lon Torrent و Solexa بود.
- روش توالی یابی lon torrent semiconductor sequencing
این روش که با نام Post_light sequencing هم معرفی میشود. در سال ۲۰۱۰ توسط شرکت Technologie Life ارائه شد. مراحل آمادهسازی و تعیین توالی این تکنیک به Pyrosequencing بسیار مشابه میباشد و از emPCR برای تکثیر قطعات استفاده میشود.
در این روش یک سیستم شناسایی نیمهرسانا مورد استفاده قرار میگیرد.همچنین تعیین توالییابی برخلاف روشهای نوری یعنی از نیروهای فلورسنت و یا لومینسانس که در سایر سیستمها مورد استفاده قرار میگیرد استفاده نمیشود؛ بلکه از شناسایی یون هیدروژن که در هنگام سنتز DNA آزاد میشود استفاده میکنند. اگر نوکلئوتیدی که به محیط وارد میشود مکمل نوکلئوتیدهای DNA هدف باشد یک یون هیدروژن آزاد میشود؛
در نتیجه یک واکنش در حسگر فوق حساس یونی رخ میدهد. در واقع از تغییرات یون هیدروژن ناشی از رها شدن یونهای مثبت هنگام سنتز رشته DNA برای تشخیص این نوکلئوتیدها استفاده میشود. این تغییرات که در زیر چاهکها قرار دارد شناسایی شده و به صورت تغییرات ولتاژ ثبت میشود. در نتیجه این تغییرات به صورت یک پیک در نمایشگر ظاهر خواهدشد.
از آنجا که این روش نیز در هر مرحله نوکلئوتیدهای مشابه و مشخص به مخلوط واکنش اضافه میکند میتوان بر اساس تولید این پیک توالی DNA را حین سنتز به دست آورد. این روش با حذف نیاز به مواد فلورسنت و دوربین سبب افزایش سرعت توالییابی و کاهش هزینهها شد. همچنین بعدها دومین دستگاه توالییابی این شرکت با نام Ion proton در سال ۲۰۱۲ عرضه شد که میزان خروجی آن بسیار چشمگیر بود
- روش توالی یابی Illumina
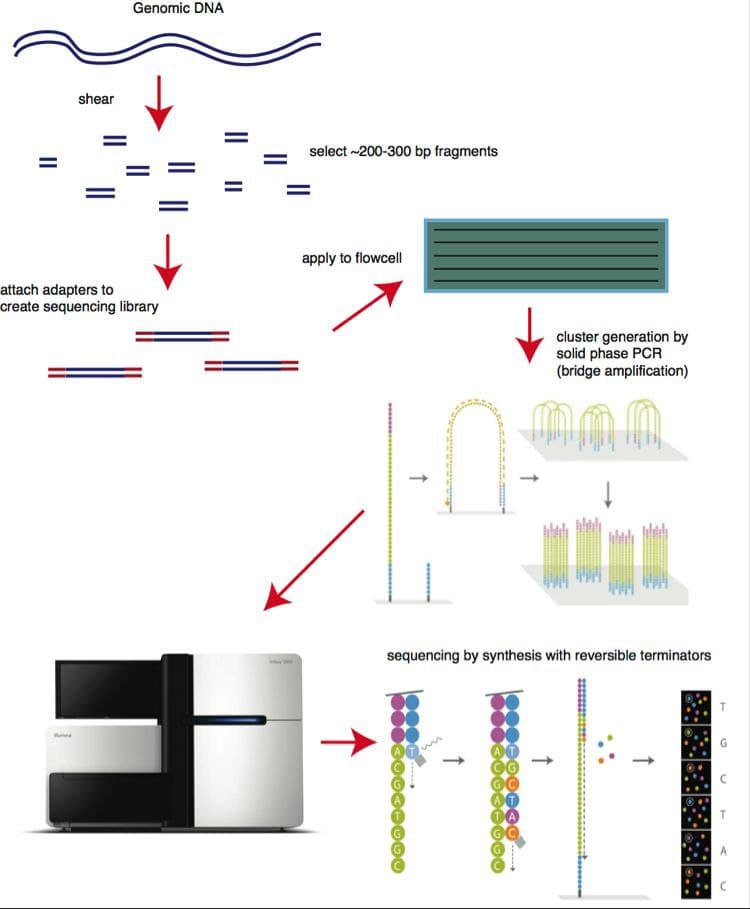
این تکنیک در سال ۲۰۰۷ ارائه شد که به جای تکثیر به واسطه بید و emPCR یا bead-based emPCR از مولکول آداپتوری استفاده شدهاست که روی یک سطح جامد ثابت شده بودند. روند کار به این صورت بود که در ابتدا ژنوم مورد نظر به قطعات ۱۰۰ تا ۱۵۰ جفت باز شکسته میشود. سپس به دو انتهای حرکت آداپتور متصل میشود و قطعات تکرشتهای میشوند. قطعات به صورت تصادفی روی سطح جامدی که با توالیهای مکمل آداپتور پوشیده شده است توزیع میشود و در نتیجه هر قطعه DNA بر روی سطح جامد ثابت میشود. در مرحله بعد انتهای آزاد این قطعات به توالیهای مکمل آدابتور متصل شده و یک پل تشکیل میدهد که پس از تکثیر این پل با افزودن مواد مورد نیاز، PCR آغاز میشود. پس از پایان هر سیکل DNA دو رشته حاصل دناتوره شده تا دوباره PCR انجام گیرد.
این واکنش تا زمانی که تعداد زیادی از قطعات مورد نظر ایجاد شود تکرار میشود. هدف افزایش تعداد قطعات مورد نظر به منظور تقویت شدن سیگنالها میباشد. این تکنیک نیازمند DNA تکرشتهای به عنوان الگو میباشد. ستهایی در حدود ۱۰۰۰ کپی از DNA تکرشتهای به صورت تصادفی بر روی سطح جامد ایجاد میشوند که به هرکدام DNA Polony گفته میشود. هر پلونی حاوی یک قطعه DNA میباشد در این روش ۴۰ میلیون پلونی به طور همزمان تعیین توالی میشوند.
نسل سوم توالی یابی
نکته حائز اهمییت راجب این موضوع این است که تکنیکهای توالییابی نسل سوم روی مولکول DNA منفرد انجام میشوند و نیازی به واکنش PCR برای آماده سازی نمونه نیست. همچنین تولید قطعات در حدود ۶ الی ۸ کیلو باز میباشد که بسیار بلندتر از طول توالی خوانده شده سایر روشهای NGS است. طول بلندتر قطعات؛ کاهش زمان، هزینه و خطای خانش را در پی دارد. این تکنیکها به SMS معروف هستند و اولین بار توسط steph Quak پایهریزی شدند و در نهایت توسط شرکتها تجاری سازی شدند.
- روش توالی یابی SMRT یا single molecule real time
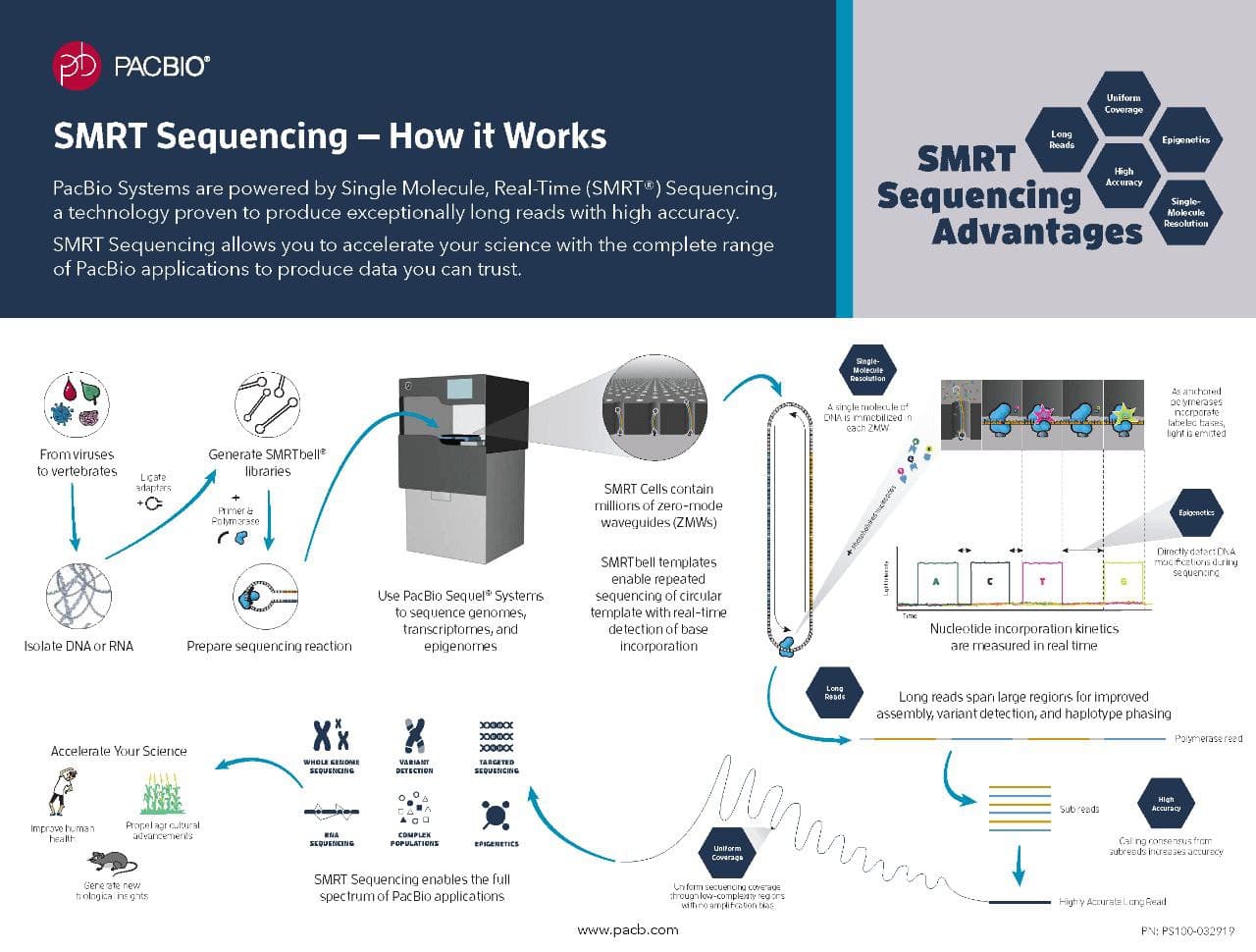
امروزه پرطرفدارترین تکنولوژی تعیین توالی نسل سوم پلتفرم شرکت Pacific Biosciences با ماشین توالییابی PacBio است. این روش بر اساس فرآیند طبیعی همانندسازی DNA طراحی شدهاست. همچنین از کارایی و درستی بالایی برخوردار میباشد. فناوری SMRT مشاهده سنتز DNA را در همان زمان وقوع امکان پذیر میسازد. در این تکنیک از چیپتهایی استفاده میکنند که دارای یک فیلم فلزی بسیار باریک میباشد. این فیلم فلزی حاوی هزاران حفره است که به آنها ZMW یا Zero_Mode Waveguid گفته میشود. قطر ZMW دو و نیم نانومتر و حجم آن در حدود ۲۰ زیپتو لیتر میباشد.
همچنین در کف چاهک یک آنزیم DNAپلیمراز تثبیت شدهاست و پلیمریزاسیون در این ناحیه انجام میشود. این چاهکها توسط دوربینهای CCA رصد میشود. ابتدا ژنوم به قطعاتی به طول هزار و ۵۰۰ جفت باز شکسته شده و به سمت هر قطعه آداپتور متصل میشود. آداپتور که در این روش مورد استفاده قرار میگیرد تفاوت خاصی با آداپتورهای مورد استفاده در سایر روشها دارد. این آداپتورها سنجاقسری نام دارند و به قطعه مورد نظر متصل میشود.
در مرحله بعد یک پرایمر و هر کدام از آداپتور متصل میشود؛ در نهایت واکنش پلیمریزاسیون با استفاده از نوکلئوتیدهایی که هر کدام با رنگ خاص لیبل شدهاند در داخل هر یک از چاهکها انجام میشود. با اتصال نوکلئوتید نشاندار به جایگاه فعال DNAپلیمراز، رنگ فلورسنت شناسایی میشود. در این روش به جای استفاده از باز نشاندار از فسفات گامای نشاندار با مواد فلورسنت استفاده شدهاست.
در نتیجه هنگام ورود نوکلئوتید به رشتهی در حال سنتز، فلوروفور همراه پیروفسفات آزاد میشود و هیچگونه ممانعت فضایی برای طول رشته در حال ساخت ایجاد نمیکند. به همین دلیل است که افزایش طول توالی خوانده میشود و همچنین شناسایی فلوروفور آزاد شده به ناحیه بسیار کوچک در کف چاهکها محدود میشود که این کار توسط متمرکز کردن نور لیزر انجام میشود. بنابراین حضور مخلوطی از نوکلئوتیدهای نشاندار هنگام انجام واکنش بلامانع بوده و سنتز رشته جدید به صورت فرآیند پیوسته دنبال میشود. بنابراین این فرآیند میتواند مولکولهای منفرد را به صورت زمان بسیار کوتاهی تعیین توالی کند. این روش دارای مزایای ارزشمندی نسبت به سایر تکنیکها است. به این صورت که قادر به تعیین توالی قطعات بسیار طولانی بیشتر از ده کیلو باز هستند. همچنین نتیجه توالی در یک روز آماده میشود امکان تجزیه و تحلیل منحصر به فرد تک مولکول در تشخیص نوع توالی خاص از یک سلول به سلول دیگر را فراهم میکند.
کاربرد توالییابی DNA
توالییابی DNA اطلاعات ژنتیکی را که در یک بخش DNA خاص، یک ژنوم کامل یا یک میکروبیوم پیچیده حمل می شود را نشان میدهد. دانشمندان میتوانند از اطلاعات حاصل از توالییابی برای تعیین اینکه کدام ژنها و دستورالعملهای تنظیم کننده در مولکول DNA وجود دارد استفاده کنند.
توالی DNA را میتوان برای ویژگیهای مشخصه ژنها، مانند خواندم قابهای باز (ORF) و جزایر CpG، غربال کرد. توالی DNA همولوگ از موجودات مختلف را میتوان برای تجزیه و تحلیل تکاملی بین گونهها یا جمعیتها مقایسه کرد. به طور قابل توجهی، توالی DNA میتواند تغییراتی در ژنی را نشان دهد که ممکن است باعث بیماری شود.
تعیین توالی DNA در پزشکی از جمله تشخیص و درمان بیماریها و مطالعات اپیدمیولوژی استفاده شدهاست. تعیین توالی قدرت انقلابی در ایمنی مواد غذایی و کشاورزی پایدار از جمله بهداشت حیوانات، گیاهان و بهداشت عمومی ایجاد کردهاست.
همچنین بهبود کشاورزی از طریق اصلاح نژاد گیاهان و دامها و کاهش خطرات ناشی از شیوع بیماری از دیگر فواید و کاربردهای آن است. علاوه بر این، توالی DNA میتواند برای محافظت و بهبود محیط طبیعی هم برای انسان و هم برای حیات وحش مورد استفاده قرار گیرد.
نتیجه گیری
تعیین توالی DNA این مکان را به دانشمندان میدهد که با مقایسه DNA بین اندامهای مختلف به ارتباط بین اندام و روابط فیزیولوژیکی آنها پی ببرند. در تعیین توالی DNA از روشهای پرتوان استفاده میشود که این امکان را فراهم کردهاست که توالیهای DNA در مدت زمان بسیار کوتاهی مورد شناسایی قرار بگیرند.
این فناوری برای بسیاری از شرکتها این امکان را به وجود آورده است تا بتوانند آن را به جایی برای آزمایشهای خانگی DNA به مشتریان خود پیشنهاد دهند. بسیاری از این آزمایشهای اختصاصی ارتباطی است که بین یک واریانت ژنتیکی و یک بیماری خاص وجود دارد. این فناوری همچنین به دانشمندان اجازه دادهاست که DNAهای بسیاری از ارگانیسمها را مورد بررسی قرار دهند تا روابط تکوینی و فیزیژنتیک بین آنها را بهتر مطالعه کنند.
گردآورنده: پریا نوذری
ویراستار: محمد پوررجب زاده
منابع:
- Heather, J. M., & Chain, B. (2016). The sequence of sequencers: the history of sequencing DNA. Genomics, 107(1), 1-8.
- Mosallayi M, Mirzaei H, Simonian M, Kheirollahi M. Next-Generation Sequencing and Applications. J Isfahan Med Sch 2016; 33(368): 2469-80
- Pourmoshir N, Mohamadi Farsani F, Vallian Borujeni S. Next generation sequencing and its application in diagnosis of genetic diseases. 3. 2017; 8 (34) :56-66
- Sabbaghi A, Soleimani M. An Overview of DNA Sequencing Methods (First Generation, Second Generation and Third Generation). Paramedical Sciences and Military Health. 2016; 11 (2) :48-60
- Mosallayi M, Mirzaei H, Simonian M, Kheirollahi M. Next-Generation Sequencing and itsApplications. J Isfahan Med Sch 2016; 33(368): 2469-80
- Pourmoshir N, Mohamadi Farsani F, Vallian Borujeni S. Next generation sequencing and its application in diagnosis of genetic diseases. 3. 2017; 8 (34) :56-66
- wikipedia.org
- https://www.cd-genomics.com/blog/dna-sequencing-definition-methods-and-applications/
- صادقی، مهدی، ۱۳۹۰، نسل جدید توالی یابی DNA، هفتمین همایش بیوتکنولوژی جمهوری اسلامی ایران، تهران، انجمن بیوتکنولوژی جمهوری اسلامی ایران
- توالییابی به روش مکسام-گیلبرت (ویکیپدیا)
- توالییابی (ویکیپدیا)

2 نظرها