
فهرست مطالب
طی دهههای گذشته روشها و تکنیکهایی در جهت درمان بیماریهای دشوار و سخت ظهور کردند که هر یک راه حلی در جهت پیشرفت علم ژندرمانی با خود به همراه داشتهاند؛ اما کاستیهای آنها از جمله کارایی کم و عدم دقت و حساسیت بالا مانع از بکارگیری آنها در مقیاس گسترده و در طولانیمدت شده که خود زمینهساز ابداع روشهای دیگری است. خالی از لطف نیست که بدانیم شروع کار ژندرمانی با استفاده از ویروسهای نوترکیب و DNA نوترکیب بوده است.
اکنون به لطف دانشاندوزیهای روزافزون بشر، اندونوکلئازهای اختصاصی نظیر “Meganuclease”، “ZFNs”، “TALENs” و در نهایت “CRISPR/Cas9” پا به صحنه گذاشتند و به محققان در ایجاد جهشهای موفق و هدفمند کمک میکنند. این امر خود جهشی بزرگ در عرصه ژندرمانی و دستکاری ژنی است که راه را برای درمان بیماریهایی نظیر سرطان و اختلالهای ژنتیکی هموار میکند. حال با توجه به تکنیکها و روشهای کشفشدهی در بازهی 2010-1966، به توضیح هریک از آنها بهاجمال میپردازیم.
حاملها در ژندرمانی
تاریخچه استفاده از حاملها به سالهای 1966 تا 1980 برمیگردد که در این بازه بحث بهکارگیری ویروسهای نوترکیب و DNA نوترکیب به میان آمد؛ اما معرفی این ابزارها به عنوان ابزاری در علم ژندرمانی، در سالهای 1983 تا 1999 انجام شد.
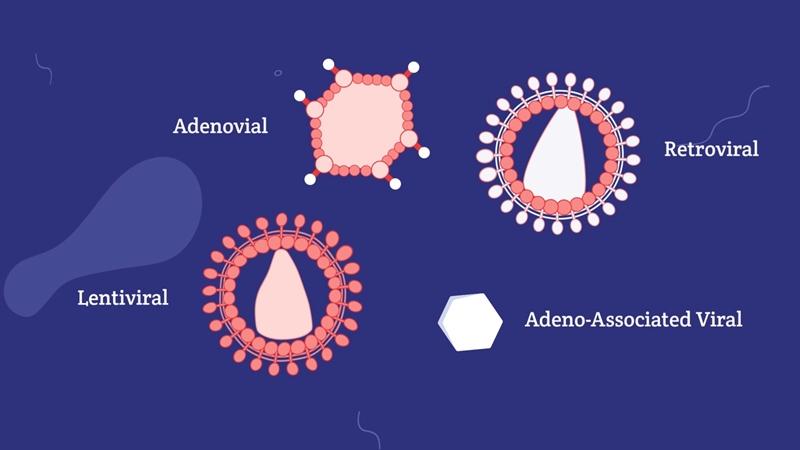
حاملهایی از جملهی حاملهای نوترکیب رتروویروسی، آدنوویروس، ویروس وابسته به آدنو (AAV)، ویروس هرپس سیمپلکس (HSV-1) و نسل اول حاملهای لیپوزومی. از سال 2000 به بعد نیز حاملهای نوترکیب غیرویروسی مانند پلاسمیدهای سنتتیک (همچون کروموزومهای مصنوعی مخمری (YAC)، باکتریایی (BAC)، پستانداران (MAC) و انسانی (HAC)) و عوامل بیوشیمایی نظیر لیپیدهای کاتیونی و پلیمرهای کاتیونی انتقالدهنده ژن، استفاده از پپتیدهای نفوذکننده به سلول (CPPs)، بهرهگیری از توالیهای الیگونوکلئوتیدی برای ژن درمانی نظیر ریبوزیمهای دارویی، عوامل مداخلهگری (RNAi) RNA، آنتیسنسها، آپتامرها و نانوذرههای دندروزومی را شاهد هستیم.
مانعی که در استفاده از این حاملها بر سر راه محققان قرار داشت، عدم دقت و هدفمندی مناسب بود که متعاقبا باعث ایجاد بیماریهایی از جمله سرطان و تحریکات ایمنی نابهجا میشد. اما با گذر زمان و با گسترش تحقیقات در حوزه حاملها و بهبود دانش در زمینه فرایندهای زیستی در انواع سلولها، حاملهایی با دقت و بهرهوری بهتر ساخته و طراحی شد. که در مطالب بعدی مجموعه اتحاد زیستشناسان ایران، به طور جامع حول مبحث حاملها توضیح داده خواهد شد.
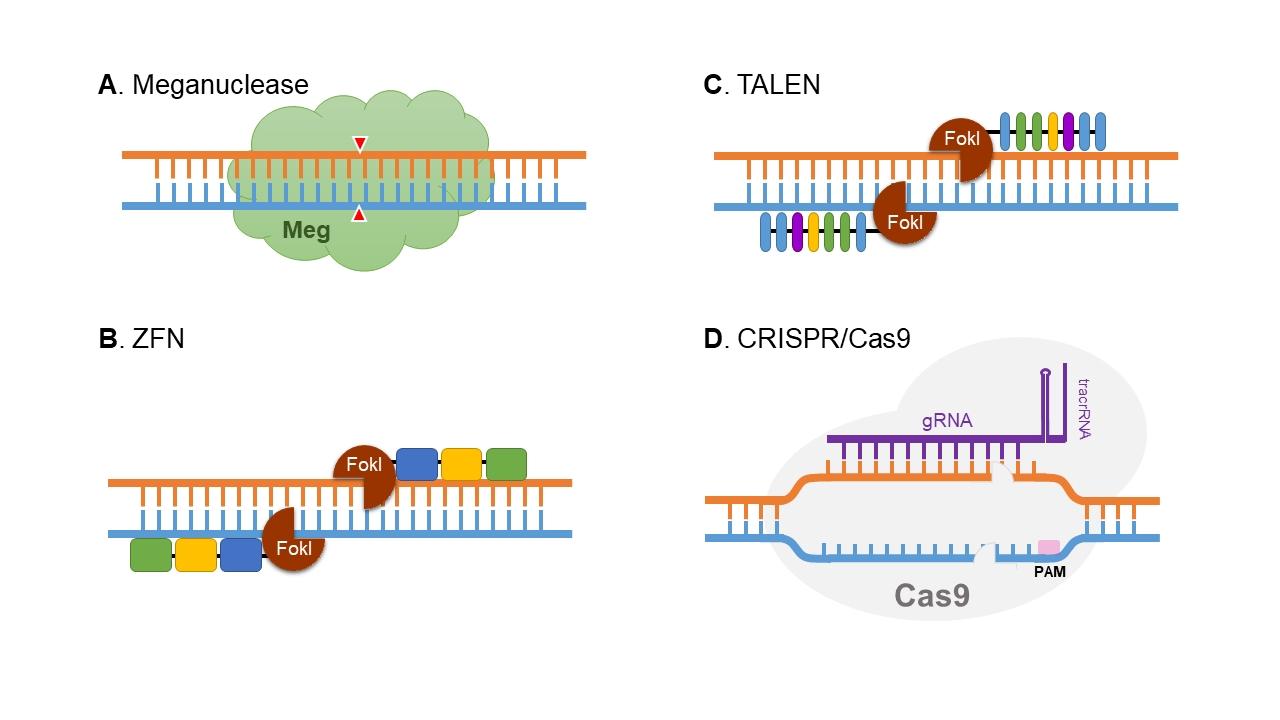
ابزارهای ویرایش ژنوم؛ DNA اندونوکلئازها
-
مگانوکلئازها (Meganucleases)
مگانوکلئازها ابتدا در مخمر، در سال 1990، شناسایی شدند و با گسترش و توسعه دانش در علم مهندسی ژن، 5 خانواده از آنها نیز کشف شده. بیشترین تحقیق و تمرکز بر روی خانواده “LAGIDADG” صورت گرفته است؛ چراکه این خانواده در تمامی موجودات وجود دارد. این مگانوکلئازها اغلب در نواحی اینترون و اینتینها (intein) وجود دارند و توسط آنها نیز رمز میشوند.
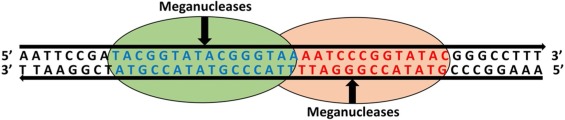
خانواده LAGIDADG میتوانند توالی به طول 40-14 نوکلئوتید را شناسایی کنند که توالی نسبتا طولانی است. این ویژگی، باعث معرفی آنها به عنوان ابزاری با کارایی و دقت بالا و همچنین اثرات مخرب کم شدهاست. با این وجود، عملکرد و وظیفه بهخصوصی در میزبان برای این پروتئینها یافت نشده است و بیشتر میل دارند در گروهی تحت عنوان «عناصر ژنتیکی خودخواه» دستهبندی شوند؛ اما استثناً دارای دو نقش اصلی در سلول هستند:
- فعالیت به عنوان یک DNA maturase
- فعالیت به عنوان یک اندونوکلئاز ویژه جهت شناسایی و شکاف توالی اگزون-اگزون (در جایی که توالی اینترون ثابت است). به دلیل همین ویژگی است که این مگانوکلئاز را «هدفیاب» مینامند.
-
نوکلئازهای انگشت روی (ZFNs)
تکنیک دقیق و هدفمند ZFNs در سال 1998 کشف شد. این تکنیک به عنوان یکی از ابزارهای مهم ژندرمانی وارد این عرصه شد. این ابزار از دو بخش پروتئین انگشتِ روی و یک آنزیم نوکلئازی تشکیل شدهاست. پروتئینهای انگشت روی با اتصال به یک نوکلئاز، کمپلکسی را تشکیل میدهند که درنتیجه میتوانند برشهایی را در دو رشتهی DNA ایجاد کنند.
پروتئینهای انگشت روی، نواحی را که باید مورد هدف قرار گیرند را شناسایی میکنند. هر پروتئین انگشت روی قادر به شناسایی 4-3 توالی نوکلئوتیدی است. دمینهای انگشت روی که خود حاوی 30 آمینواسید است، به یک اتم روی متصل شدهاند؛ این اتصال از طریق اسیدهای آمینه محافظتشده Cys2His2 صورت میگیرد. این پروتئینها ساختاری پیچیده و متراکم را تشکیل میدهند که دارای یک مارپیچ آلفا و دو صفحه بتا است که همین مارپیچ آلفا سبب ایجاد اتصالاتی در توالی DNA (در محل شکاف بزرگ) میشود.
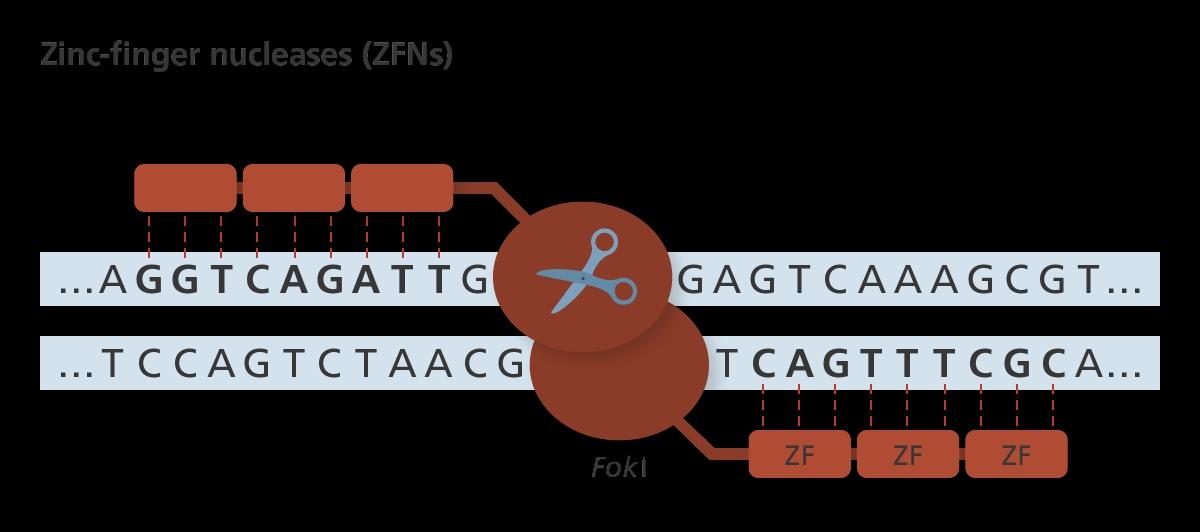
نوکلئاز معمول در این تکنیک، FolkI نام دارد که برای متصلشدن به DNA باید حتما به صورت دایمر درآید. دایمر در FolkI متشکل از دو موتیف پروتئینی است. یکی از این موتیفها به DNA به صورت اختصاصی متصل میشود و دیگری در پایانه کربوکسیلی DNA فعالیت اندونوکلئازی را در دست میگیرید. عدم اتصال دقیق این انگشت رویها (ZF) به سه جفت بازی که برای آنها طراحی شده، از چالشهای این تکنیک است. همچنین برای اتصال به تمامی نواحی مورد نظر در DNA ما به 64 جفت موتیف انگشت روی نیازمندیم که در حاضر چنین مجموعهای از این موتیفها در دسترس نیست؛ اگرچه تکنیکهای کامپیوتری در ساخت موتیفها کمککنندهاند اما امکان طراحی همه آنها وجود ندارد.
-
نوکلئازهای اثرکننده شبهفعالکننده رونویسی (TALENs)
تکنیک TALENs که تحولی بزرگ در علم مهندسی ژن ایجاد کرد در سال 2009 و به سرعت پس از ZFN کشف شد. پروتئینهای TALENs (Transcripion Activator-Like Effectors Nucleases) به اثرگرهای شبهفعالکننده شناخته شدهاند. این پروتئینها از باکتری زانتاموناس (Xanthomonas spp) که یک باکتری بیماریزای گیاهی است گرفته شده و با DNA گیاه برقرار میکنند.
TALENs از دو جز فاکتورهای شبه فعالکننده ترجمه (در انتهای N قرار دارد) و دمین متصلشونده به DNA (در انتهای C قرار دارد) تشکیل شده است. در اینجا همانند روش ZFN ما شاهد فعالیت اندونوکلئاز FokI هستیم که برای برش DNA یا DSB باید به شکل دایمر درآید. در اینجا منظور از فاکتورهای شبهفعالکننده رونویسی، TALENs و دمین متصلشونده به DNA، اندونوکلئاز FokI است. در این سیستم هر TALENs از 2 توالی تکراری (یکی در رشته بالا و دیگری در رشته پایینی DNA) به طول 20-15 عددی به نام RVDs تشکیل شده که هر RVD یک تک نوکلوتید را از DNA شناسایی میکند.
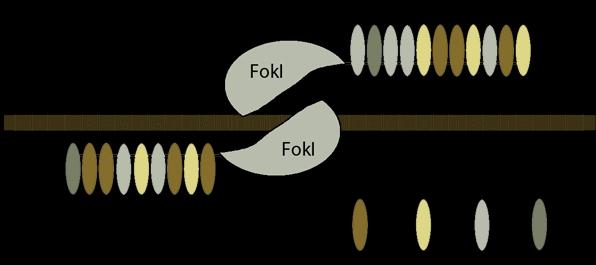
روش TALENs کارایی و بازده بیشتری نسبت به ZFNدارد؛ اما به دلیل اینکه توالیهای TALEN تکراری و شبیه به هم هستند، کار را در مراحل جمعآوری این توالیهای تکراری و بارگذاری در یک پلازمید و در نهایت کدکردن آنها در یک سلول میزبان سخت میکند. به جبران این عیب، از اندونوکلئازهای نوع II استفاده کردند که کتابخانههای پلازمیدی TALE را برش میزد و به دنبال آن چندین توالی TALE را به هم متصل میکرد. با این کار دیگر محدودیتی در مورد هدف قراردادن توالی ژنومی مورد نظر وجود نداشت. این روش با استفاده از شیوه کلونیگ استاندارد به روش “Golden Gate” صورت میگیرد. با این شیوهی طراحی توالی TALE، امکان ساخت انواع فاکتورهای شبه فعالکننده رونویسی برای مهندسی ژنوم وجود دارد.
-
سیستم نوین CRISPR-Cas9
تکنیک کریسپر (Regularly Clustered Palindromic short Interspaced Repeats) برای اولینبار توسط محققان ژاپنی در سال 1987 کشف شد. کریسپر درواقع سیستم ایمنی باکتریایی است که از باکتری در برابر فاکتورهای بیماریزا همچون پلازمیدها و یا فازها محافظت میکند. این سیستم ابتدا در باکتری اشرسیا کلی (E.coli) کشف شد. کریسپر با حفظ و نگهداری توالیهای مهاجم و به اصطلاح با ایجاد یک حافظه بیولوژیک از خود در برابر عوامل بیگانه محافظت میکند.

در سال 1987 تنها به این کشف رسیدند که 29 توالی تکراری در بین 32 توالی غیرتکراری در ژنوم باکتری جای گرفته است. در سال 2000 توسط موجیکا و دیگر محققان، مشخص شد که سیستم کریسپر در 40 درصد باکتریها و 90 درصد آرکیها وجود دارند. این زمان اولینباری بود که از واژه CRISPR برای معرفی این سیستم بهکار برده شد. سرانجام در سال 2002 مشخص شد که CRISPR سیستم ایمنی باکتریایی است که ویروس باکتریها (باکتریوفاژها) توانایی آلودهکردن و از بین بردن باکتریها را ندارند.
در سال 2011 بود که بهطور جامع و دقیق اجزا، کارکرد، انواع سیستم کریسپری و معایب و مزایای آن به طور کامل ارائه شد. سیستمهای ویرایش ژنومی با اصلاح، حذف یا قراردادن یک توالی سالم از ژن مورد نظر، ما را در درمان یا کمکردن شدت بیماریها تجهیز کردهاند. انتظار داریم با پیشرفت روزافزون علم و خلق روشها و دستاوردهای نوین، پنجری جدیدی در درمان بسیاری از بیماریهای ژنتیکی دشوار باز شود.
سارا تاجداری
منابع:
https://www.researchgate.net/publication/315791735_CRISPR-Cas9_Structures_and_Mechanismshttps://www.ncbi.nlm.nih.gov/pmc/articles/PMC7140808/https://www.ncbi.nlm.nih.gov/pmc/articles/PMC3176093/

2 نظرها